Thème de recherche n°1 : La dégradation des ARNs chez Bacillus subtilis
Le contrôle de la demi-vie d'un ARN messager est un des mécanismes majeurs permettant à la cellule de réguler l'expression de ses gènes. Notre groupe étudie les voies de la maturation et la dégradation de l’ARN chez la bactérie modèle à Gram-positif du sol, Bacillus subtilis. La compréhension de ces mécanismes s’est fortement améliorée ces dernières années. Sur plus de trente-cinq ribonucléases bactériennes connues à ce jour, pratiquement toutes ont été identifiées que ce soit chez E. coli ou chez B. subtilis. Huit enzymes seulement sont communes aux deux bactéries. Celles-ci sont de loin les mieux conservées, leurs traces étant retrouvées au-delà même du monde procaryotique. A noter que certaines ribonucléases essentielles chez E. coli n'existent pas chez B. subtilis et vice versa. Deux voies majeures de la dégradation de l’ARNm sont actuellement connues chez B. subtilis. Dans la première, l’ARNm est clivé par une endoribonucléase, la RNase Y dans la plupart des cas. Cette enzyme est localisée dans la membrane et est considérée comme l’analogue fonctionnel de la RNase E d’E. coli. Les fragments d’ARN générés par clivage endonucléolytique sont ensuite dégradés dans le sens 3’-5’, principalement par la PNPase, et dans le sens 5’-3’ par le complexe RNase J1/J2. Cette activité exoribonucléolytique capable de digérer l’ARN dans le sens 5’-3’ n’a pas été identifiée chez E. coli, ce qui constitue une différence majeure entre ces deux organismes. Dans la deuxième voie, une RNA pyrophosphohydrolase déprotège les transcrits primaires en enlevant le groupement triphosphate de leur extrémité 5’. L’ARN monophosphorylé est ensuite dégradé jusqu’au bout par l’activité 5’-3’ exoribonucléolytique de la RNase J1/J2. Cette voie est très similaire à la voie de dégradation des ARN chez les eucaryotes catalysée par l’enzyme de ‘decapping’ et Xrn1.
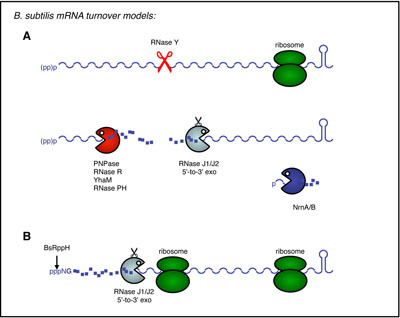
Les dernières découvertes de l'équipe ont permis de mieux comprendre la dégradation des ARNm chez B. subtilis:
